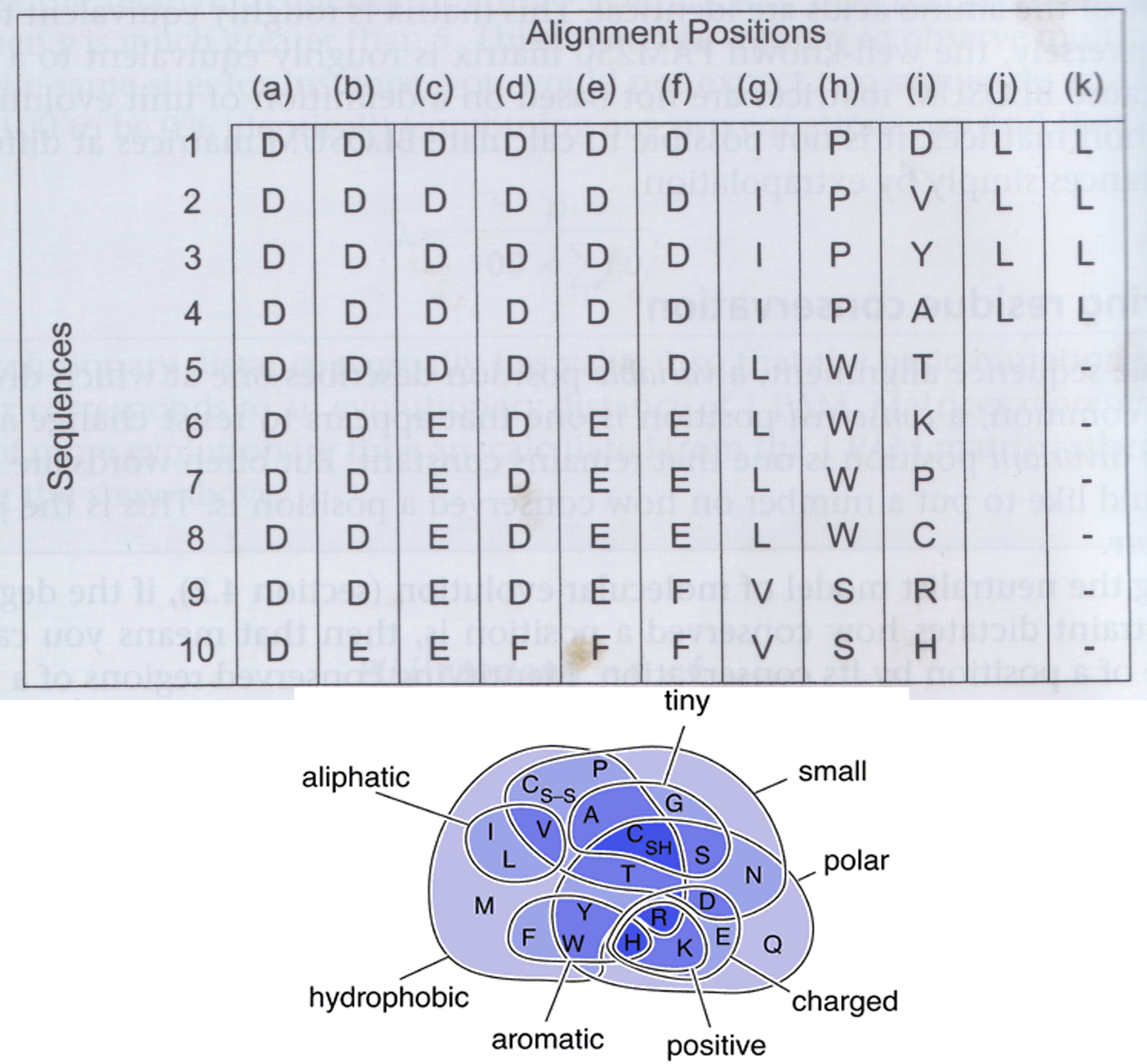
Considerar primero las columnas (a) hasta (f) que están organizadas por orden de homologia:
Accede al código una letra de los aa
- (a) es evidente que es lo más conservado de todo ya que solo contiene un aa D (Asp)
- (b) contiene un solo aa variable E (Glu)
- (c) Tiene los mismos aa que (b), pero ninguno de ellos es dominante, y es por tanto más variable.
- (d) tiene un solo aa F (Phe), pero esta posición se le considera menos conservada que las anteriores por las diferentes propiedades fisico-químicas (F es aromático e hidrofóbico, mientras que D y E son ambos polares y con carga). Una posición que tolera cambios menores (D<->E) se le considera más conservada que una que admite grandes cambios (D<->F)
- (e) implica cambios conservativos (D<->E) y no conservativos (D<->F, E<->F)
- (f) es más variable por la variación del número de aminoácidos que varían
NOTA: Es posible considerar que la columna (g) está más conservada que la (e) y la (f) ya que los 3 aa son similares
Consideremos ahora las columnas desde (g) a (i)
- Ambas columnas son equivalentes en el número y frecuencia de los aa, pero las propiedades fisico-quimicas de los aa en h son mucho más variables
- (i) es las más variable de todas las presentadas hasta ahora
Las columnas (j) y (k) ilustran la importancia de los huecos.
- (j) proviene del alineamiento de solo 4 secuencias. En este caso han coincidido 4 Leu en las 4 secuencias comparadas, indicando que dicha secuencias debe estar conservada y es importante
- Pero (k) proviene de un alineamiento con 10 secuencias, en las que en 6 de ellas hay ausencia de L (Leu). Esto indica claramente que esta Leucina es la menos conservada de todas, porque su ausencia no provoca deterioro en la funcionalidad de la proteína